科研 | Water Research:废水排放引起微生物群落转变并改变营养状态
实验设计

结果
1 检测群落转变的阈值群落WET试验确定了引起河流细菌群落明显变化的污水浓度,揭示了与废水污染相关的营养变化的潜在阈值(图1b,表S7)。具体来说,细菌群落在nMDS轴1上从高(即50%和90%)到中等(即10%)和低(即0%对照、0.1%和1%)污水浓度的梯度上有所不同(图1b)。营养物浓度(即TN、TKN、NH3、NOx、TP)和BOD在很大程度上与污水浓度呈协变关系(图1c,表S5和6),并与细菌群落的变化显示出强烈的正相关关系(图1d)。细菌群落结构的最大变化发生在中等(>10%)的污水浓度以上,表明有可能从中营养状态过渡到富营养状态。此外,细菌群落沿着污水浓度的梯度变化与参与硝酸盐还原的基因(napA/narG)呈正相关(图1e)。相反,与反硝化作用(nirS)和硫酸盐还原作用(dsrA和phsA)相关的基因与nMDS轴2呈正相关,表明与不同的污水类型有关,而不是污水浓度。现实世界的污水排放也发生了类似的细菌群落变化(图2a)。在群落水平WET测试中,观察到现场采样的群落结构在代表~1%(WWTP 3)、~10%(WWTP 1)、~50%(WWTP 4)和~90%(WWTP 2)污水浓度的污水类型中明显分离(图2b)。具体来说,TN、NOx和TKN在不同的采样点之间具有高度的异质性,在两个采样时间内显示出从高(即WWTP 2)到中等(即WWTP 1和4)和低(即WWTP 3)污水浓度的一致梯度。尽管TN和TKN有一些局部的变化,但与上游背景值相比,排污口下游的营养物浓度(特别是氮氧化物)普遍增加(图2b)。

图1. 群落WET测试-(a)整个研究河口的污水处理厂位置;(b)细菌群落的nMDS排序;(c)环境变量,(d)营养物质,和(e)基因驱动群落转变。

图2. 微生物实地调查-(a)细菌群落的nMDS排序;(b)污水处理厂排放污水中主要营养物质的平均浓度;(c)在(a)中推动群落转变的环境变量。2 营养转变的指标在群落WET测试中观察到群落优势的变化与污水浓度有关(图3)。在所有样品中,Burkholderiales(Betaproteobacteria)是最丰富的细菌目,约占序列的20%以上,而Frankiales(Actinobacteria)在丰富的细菌目中占前10%,约占序列的3%。一般而言,Burkholderiales的丰度随着污水浓度的增加而增加,而Frankiales则相反。这些差异可以用两个潜在的关键类群来解释:Malikia spp.(占7.5%;36%的OTUs属于Burkholderiales)和hgcI_clade(占2.7%;98%的OTUs属于Frankiales)。Malikia spp.受中等污水浓度(1-10%)刺激,生长稳定在较高浓度(10-90%),表明其与中营养和富营养化条件的潜在亲和力(图4,图S5a)。相比之下,hgcI_clade丰度在贫营养条件下(低污水浓度)稳定,在污水浓度高于1%时被抑制。网络分析确定了Malikia spp.和hgcI_clade、功能基因丰度和群落WET测试中环境条件之间的统计联系(图5a、b)。Malikia spp.与BOD、narG基因(硝酸盐减少)以及营养浓度(包括TN和NOx)呈强正相关,与TKN和TN:TP呈弱正相关。相比之下,hgcI_clade与营养浓度(包括TN、TKN和NOx)、TN:TP、narG和napA基因(硝酸盐减少)呈强负相关,并与phoD基因(产生生物可利用磷)、nirS基因(反硝化)和dsrA基因(硫酸盐还原)呈弱正相关。此外,Malikia spp.和hgcI_clade与Burkholderiales、Rhodobacterales、Pseudomonadales和Sphingobacteriales的其他不太丰富的属有密切的联系,这为微生物组内的潜在相互作用提供了一些额外的见解。
特别声明:以上内容(如有图片或视频亦包括在内)为自媒体平台“明珠号”用户上传并发布,本平台仅提供信息存储服务。
-
 大 空腹喝牛奶会拉肚子?关于喝牛奶的3个误区!
大 空腹喝牛奶会拉肚子?关于喝牛奶的3个误区! -
 人 转告家人:中老年人想要拥有好睡眠,睡前尽量不要做这5件事
人 转告家人:中老年人想要拥有好睡眠,睡前尽量不要做这5件事 -
 极 中秋节后的月饼去哪儿了?“按销定产”延长销售周期配合促销打折,过期月饼就销毁
极 中秋节后的月饼去哪儿了?“按销定产”延长销售周期配合促销打折,过期月饼就销毁 -
 穴 颈椎病、肩周炎、落枕,按按手臂上一条线,全解决
穴 颈椎病、肩周炎、落枕,按按手臂上一条线,全解决 -
 上 中药材掀“涨价潮”!返乡大学生回家“种药”,年入超40万元
上 中药材掀“涨价潮”!返乡大学生回家“种药”,年入超40万元 -
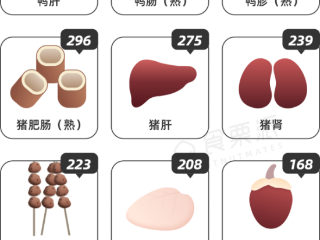 e 这些肉,升尿酸比海鲜还猛
e 这些肉,升尿酸比海鲜还猛 -
 潇 好不容易准备结婚,杭州女孩却查出“接吻病”!医生提醒:这类人要小心!
潇 好不容易准备结婚,杭州女孩却查出“接吻病”!医生提醒:这类人要小心! -
 时 新冠疫苗成“印钞机”,科兴半年大赚500亿元,或赶超辉瑞登顶榜首
时 新冠疫苗成“印钞机”,科兴半年大赚500亿元,或赶超辉瑞登顶榜首 -
 茶 什么时辰喝什么茶,不可不知的喝茶学问!
茶 什么时辰喝什么茶,不可不知的喝茶学问! -
 八 14岁学生死于炭疽,北京、山东、山西多地报告疫情,这条传播链值得警惕
八 14岁学生死于炭疽,北京、山东、山西多地报告疫情,这条传播链值得警惕 -
 那 天然“促睾”食物被找到,男性若想提高活力,请舍得吃
那 天然“促睾”食物被找到,男性若想提高活力,请舍得吃 -
 腾 生过孩子的女人,身体有啥“变化”?这4个不同,很明显!
腾 生过孩子的女人,身体有啥“变化”?这4个不同,很明显!









































